Revista Científica UDO Agrícola Volumen 9.
Número 4. Año 2009. Páginas: 837-847
Caracterización
mediante polimorfismo del ADN amplificado al azar de aislamientos de Colletotrichum gloeosporioides Penz.
obtenidos en frutos de aguacate (Persea
americana Mill.) cv. Hass procedentes de zonas agroecológicas de Michoacán,
México
Characterization of Colletotrichum gloeosporioides Penz. isolates obtained from avocado
(Persea americana Mill.) fruits cv. Hass from different
producing regions of Michoacán, Mexico using Random Amplified Polymorphic DNA
José Luciano MORALES GARCÍA![]() 1, María del Pilar RODRÍGUEZ GUZMÁN2,
Hilda Susana AZPÍROZ RIVERO3 y Martha Elena PEDRAZA SANTOS1
1, María del Pilar RODRÍGUEZ GUZMÁN2,
Hilda Susana AZPÍROZ RIVERO3 y Martha Elena PEDRAZA SANTOS1
1Facultad
de Agrobiología “Presidente Juárez” Universidad Michoacana de San Nicolás de
Hidalgo, México, 2Instituto de Fitosanidad, Colegio de
Postgraduados. Especialidad en Fitopatología. 56230, Montecillo, Texcoco,
México y 3Instituto Nacional de Investigaciones Forestales,
Agricolas y Pecuarias (INIFAP). Campo Experimental Valle de México. km 18,5
Carretera Los Reyes-Lechería, Chapingo, Texcoco, México. E-mails: jluciano@umich.mx y
jluciano@prodigy.net.mx ![]() Autor para
correspondencia
Autor para
correspondencia
|
Recibido: 31/01/2009 |
Fin de primer arbitraje:
20/03/2009 |
|
Primera revisión recibida: 20/08/2009 |
Aceptado: 21/08/2009 |
RESUMEN
El
síntoma de antracnosis del aguacate, causada por Colletotrichum gloeosporioides, aparece con variaciones en los
municipios de Michoacán. El objetivo de la presente investigación fue
caracterizar aislamientos monoconidiales de C.
gloeosporioides, obtenidos de frutos de aguacate con síntomas diversos de
antracnosis y procedentes de diferentes regiones productoras de Michoacán, utilizando sus huellas
genómicas producidas por la técnica de
reacción en cadena de la polimerasa (PCR) a través del polimorfismo del ADN
amplificado al azar (RAPD). La distancia genética más pequeña se observó entre
los monoconidiales 30 y 33 de la localidad de Uruapan con síntomas de
antracnosis y antracnosis con coarteadura. Las distancias más grandes se
observaron entre 11 y 17 procedentes de Tacámbaro y producen síntomas de
antracnosis con coarteaduras y viruela
respectivamente. El análisis de agrupamiento muestra grupos que se forman con
base a localidades y síntomas.
Palabras claves:
Aguacate, antracnosis, Colletotrichum,
RAPD, PCR.
ABSTRACT
Avocado anthracnose by C. gloeosporioides is present under
several variations in all the counties of Michoacan, Mexico where avocado is
cultivated. The objective of the present research was to characterize
monoconidial isolations of C. gloeosporioides,
obtained from avocado fruits with different symptoms and coming from different
producing regions of Michoacan, using their genomic tracks produced by the
polymerization chain reaction technique through the random amplified
polymorphism of ADN (RAPD). The
smallest genetic distance was observed among the monoconidial isolates 30 and
33 from Uruapan fruit with anthracnoses and crack-anthracnoses symptom. The longest distances were observed
among the isolates 11 and 17 are from Tacámbaro producing crack-anthracnoses
and smallpox symptoms respectively. Group seems to indicate that groups are
formed based on the localities and
symptoms.
Key words: Avocado, anthracnoses, Colletotrichum, RAPD, PCR.
INTRODUCTION
México es el principal productor de aguacate (Persea americana Mill.) en el mundo,
aportando cerca de 1.100.000 t en aproximadamente 124.829 ha, que corresponden
al 52% de la producción mundial. Michoacán participa con 84.6% de 753.801 ton producidas a nivel nacional de las cuales exportó 42.307 ton en
1998-1999.
Entre los factores fitopatológicos que más limitan la
exportación, se encuentran la antracnosis y roña (USDA, 1997; Lázaro, 1985 y
Téliz, 1999). Colletotrichum
gloeosporioides Penz. causa la antracnosis en una amplia gama de
hospedantes. Se han reportado severas pérdidas en cultivos como: aguacate,
berenjena, uva, mango, entre otros (Mills et
al., 1992). La antracnosis se encuentra distribuida en todos los municipios
donde se cultiva aguacate, el hongo afecta al fruto en cualquier etapa de su
desarrollo, traslado, almacenaje y comercialización. Una humedad relativa de 85-90%, temperatura
de 18-25 °C en campo, y la falta de aereación en los huertos favorecen la
infección. El hongo sobrevive en brotes tiernos, frutos momificados, ramas y
hojas afectadas. La infección más severa se presenta durante los meses de
junio, julio y agosto que es cuando se presenta la época de lluvias y en menor
grado en febrero y marzo cuando se presentan lluvias ocasionales llamadas
“cabañuelas”.
Los municipios con mayor incidencia son: Tacámbaro
74%, Tingüindín 67%, Uruapan 57%, Peribán 50% y Ziracuaretiro 42 % (Morales, 1996). La disponibilidad de un método confiable,
sensitivo y rápido para la detección temprana de C. gloeosporioides podría facilitar los estudios epidemiológicos y
la implementación de medidas de control apropiadas; sin embargo, los síntomas
de la enfermedad en hoja son difíciles de diagnosticar, necesitándose la
identificación del patógeno mediante características culturales y microscópicas
de aislamientos de tejido enfermo (Mills
et al., 1992). La caracterización
molecular para diferenciar aislamientos monoconidiales de C. gloeosporioides sobre la base de sus huellas genómicas, es una
herramienta adicional que ayuda a su identificación precisa, basada en un análisis directo del ADN, dando así
algunas ventajas sobre los métodos tradicionales, que se ven influenciados por
las condiciones ambientales. En la década de los 90’s la amplificación del ADN
mediante la técnica de
La diversidad en el tipo de síntomas que C. gloeosporioides causa en el fruto de aguacate como; antracnosis,
antracnosis con coarteaduras, viruela y manchas superficiales puede deberse a
la variación genética del hongo o a la influencia de las condiciones climáticas. El uso de técnicas
como la de RAPD permite un análisis preciso y confiable de las variaciones
genéticas del patógeno. Los diferentes aislamientos de C. gloeosporioides poseen
secuencias de ADN diferentes por lo que el uso de iniciadores decámeros puede
producir huellas genómicas diferenciales por medio de RAPD y con ello permitir
estudios de caracterización de germoplasma y detección de variabilidad genética
(Bailey y Jeger, 1992). La caracterización e identificación de hongos
fitopatógenos se ha basado tradicionalmente
en características morfológicas, culturales, y patogénicas. Debe considerarse
que estas técnicas no siempre son confiables debido a que el ambiente influye en su manifestación; sin
embargo, recientemente se han desarrollado otros métodos complementarios de laboratorio con resultados muy
prometedores, uno de ellos es la identificación
de isoenzimas específicas (Morales, 1996). Otras son las técnicas moleculares basadas en el manejo
del ADN, como la amplificación de fragmentos por medio de
El análisis genético de poblaciones necesita de
marcadores genéticos que den clara información. Los patólogos han empleado
aquellos que son relevantes en la agricultura y fácilmente disponibles, como la
virulencia de los patógenos sobre un juego de plantas que contienen diferente
resistencia genética o a fungicidas específicos. La relación entre fenotipos y
genotipos caracterizada mediante marcadores, proporciona información sobre la
estructura genética de los patógenos (Kolmer, 1991). Aunque las características
de virulencia y sensibilidad a fungicidas
no son de mucho peso, dan bases
estimativas de la potencialidad genética en poblaciones de patógenos
(McDonald y McDermott, 1993).
El objetivo planteado en la presente
investigación fue la caracterización y
comparación de la diversidad genética, síntomas y origen de aislamientos de C. gloeosporioides mediante la técnica
de RAPD.
MATERIALES Y MÉTODOS
Localización del área de estudio
La presente investigación comprendió dos fases, una de campo y otra de
laboratorio. La fase de campo consistió en hacer un muestreo dirigido en
huertos de diferentes localidades de tres zonas agroecológicas para colectar
frutos de aguacate cv. Hass con alguno de los síntomas producidos por C. gloeosporioides. La fase de laboratorio se realizó en el
laboratorio de frutipatología del Colegio de Postgraduados en Montecillo Edo.
de México y el Laboratorio de Marcadores Genéticos Moleculares ubicado en el
Campo Experimental Valle de México (CEVAMEX) perteneciente al Instituto
Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP).
Colecta de material
Se colectaron frutos de aguacate cv. Hass con síntomas de antracnosis, antracnosis con
cuarteaduras, manchas brillantes, y viruela, de diferentes zonas agroecológicas
(Cuadro 1). Las muestras en bolsas de
plástico se trasladaron al laboratorio de frutipatología del Colegio de
Postgraduados.
|
Cuadro 1. Clase numérica de monoconidiales de Colletotrichum gloeosporioides aislados
de frutos de aguacate cv. Hass, síntomas y lugar de aislamiento en el estado
de Michoacán en 1996. |
||||||
|
|
|
|
Síntomas de
monoconidiales |
|||
|
Región |
Localidad |
msnm |
A |
AC |
V |
MS |
|
Uruapan |
Uruapan |
1675 |
|
1 |
|
42, 47, 48, 49 50, 51, 52 |
|
Jucutacato |
1620 |
|
|
4, 5 |
|
|
|
Toreo el Alto |
1650 |
45 |
|
6, 7, 8 |
|
|
|
Tingambato |
1780 |
|
|
32 |
46 |
|
|
La col |
1350 |
|
31 |
3 |
|
|
|
La mesa |
1300 |
|
|
9 |
|
|
|
San Juan Nuevo |
|
30 |
34 |
|
|
|
|
El Durazno |
2270 |
2 |
|
35 |
|
|
|
Comunal |
2300 |
5 |
|
|
|
|
|
Panguaro |
|
|
39 |
41 |
|
|
|
Milpillas |
|
4 |
33 |
|
|
|
|
Taxuri |
1695 |
|
40 |
|
|
|
|
Meson |
1600 |
|
43 |
|
|
|
|
El cuate |
2000 |
|
|
36, 37 |
26, 29 |
|
|
N. Zirosto |
1975 |
|
25 |
|
|
|
|
S. Francisco |
1635 |
|
|
27, 28, 53 |
|
|
|
Tacámbaro |
Tacámbaro |
1930 |
|
|
22, 27 |
|
|
las Joyas |
|
|
|
20 |
|
|
|
Viñas |
|
|
|
21 |
|
|
|
Ario de Rosales |
|
|
|
10 |
|
|
|
Longaniza |
|
15 |
|
|
|
|
|
Potrero |
|
44 |
|
36 |
|
|
|
Tarascón |
2220 |
13, 14 |
11 |
16 |
19 |
|
|
La Españita |
|
|
|
17 |
|
|
|
Jujucato |
|
|
|
|
12 |
|
|
MC = Monoconidial; A = Antracnosis; AC = Antracnosis con coarteaduras; V = Viruela;
MS = Manchas Superficiales |
||||||
Fase de laboratorio
Las muestras se llevaron al laboratorio donde se procedió a la siembra de tejidos con síntomas
de antracnosis en medio de cultivo PDA. Dos días después se reaislaron los
diferentes hongos con la finalidad de purificarlos. En la medida que los hongos
llenaron las cajas Petri se hicieron preparaciones temporales con lactofenol
azul, para posteriormente observarse al microscopio compuesto. Cuando estos
aislamientos esporularon se procedió a su
identificación mediante el uso de claves especializadas. De esta manera
se lograron aislamientos puros de Colletotrichum
gloeosporioides, y se procedió a
obtener cultivos monoconidiales. Según el método propuesto por Morales (1996).
Para la caracterización con RAPD´s se usaron 20 monoconidiales los cuales
presentan diferentes síntomas de la enfermedad y provienen de las regiones de
Tacámbaro y Uruapan. Para tener
suficiente micelio y realizar la extracción de ADN, se colocaron tres rodajas
de
El micelio se colocó dentro de una pequeña bolsa de
malla de fibra de vidrio. Las muestras
se colocaron en una hielera de unicel y se congeló con nitrógeno líquido para
detener los procesos fisiológicos y bioquímicos sin causar daño y se
almacenaron a - 80°C para posteriormente liofilizarlos en un equipo Labconco a
– 51 ºC y un vacío de 006 vacuum x10-3 mbar durante 48 horas con la
finalidad de deshidratar los tejidos por sublimación. La cámara se mantuvo a
una temperatura de - 55 °C con un vacío £ 100 micrones. El micelio liofilizado se molió con un
molino de café durante 10 seg. y el
polvo fino (a mayor finura mayor obtención de ADN) se almacenó en tubos corning
de 15 ml en el refrigerador (±
Cada
reacción de 25 µl contenía: ADN, iniciador, un amortiguador, los cuatro
nucleótidos, cloruro de magnesio, Taq polimerasa y agua bajo las
concentraciones indicadas en el Cuadro 2.
|
Cuadro 2. Componentes y concentraciones de los
componentes necesarios para efectuar una reacción de amplificación mediante
la técnica de RAPD. |
|
Componentes
|
Concentración Final |
|
Templado de ADN (30 ng/µl) |
100
ng |
Iniciador (10µM)
|
1,5 µM |
|
Buffer PCR 10 X, libre de
magnesio |
1
X |
Taq polimerasa (5 u/µl)
|
1,5
u |
|
dNTPs (200 µM) |
2,0
µM |
|
MgCl 2 ( |
2,5
mM |
|
ddH2O |
6,8
µl |
En cada uno de los tubos se colocaron 3,33 µl del templado (ADN del
monoconidial) + 21,7 de la mezcla de reacción. Posteriormente se cubrió la
solución de cada tubo con 25 µl de aceite mineral estéril. Se colocaron los
tubos eppendorf con la muestra en el termociclador (TECHNE PHC-2) y se corrió
el siguiente programa: 1 ciclo de 7 min a 94 °C, 45 ciclos compuestos de 3
segmentos como sigue: 1,5 min. a 94 °C, 2
min. a
Análisis de los productos amplificados:
Preparación del gel
Electroforesis. Los
productos amplificados se separaron en un
gel de agarosa al 1.4 % de 14 x 9 cm, el cual se colocó dentro de la
cámara de electroforesis, se llenó con
amortiguador TAE IX (Trisma base, acetato de sodio y EDTA) hasta que el gel estuvo aproximadamente a
Análisis
estadístico de la variabilidad genética
Con los datos
obtenidos de la identificación de cada banda por monoconidial seleccionadas en
base a la claridad y nitidez de los productos amplificados, se codificó en
código binario para calcular las distancias genéticas entre las cepas, mediante el programa
RAPD-104 usando el estimador de
Rogers (Armstrong et al., 1995).
Después de calcular las distancias genéticas se procedió a hacer el análisis de
agrupamiento, mediante un análisis multivariado con agrupamiento completo según
los métodos de SAS (1988).
Representación gráfica. Se realizó un
análisis de componentes principales mediante los métodos propuestos por SAS
(1988), con la finalidad de observar en el espacio la distribución de la
composición genética de los monoconidiales estudiados en función de las
localidades y del síntoma. Cálculo del índice de variabilidad genética. Con la matriz
de presencia/ausencia de bandas se calculó el índice de variabilidad genética
propuesto por Montero et al., (1998).
RESULTADOS Y DISCUSIÓN
La fase de caracterización de los 20
monoconidiales MC3, MC6, MC7, MC8, MC9,
MC10, MC11, MC12, MC13, MC15, MC17, MC21, MC30,
MC33, MC37, MC40, MC42, MC44, MC45 y MC46 mediante RAPD se realizó
usando los iniciadores OPM-10, OPL-12, OPI-16, OPD-11 y OPA-16. El patrón de
bandas obtenido por cada iniciador para cada
una de las cepas se observa en las figuras 1 al 5. Es importante hacer
notar que con cuatro de los iniciadores algunas de las cepas no amplificaron
ninguna banda, lo cual podría explicarse de dos formas: 1) que el iniciador no
haya encontrado ninguna región homologa
con el ADN genómico del hongo, 2) que si haya existido alguna región homologa,
pero las condiciones de amplificación no fueron suficientes para amplificarlo,
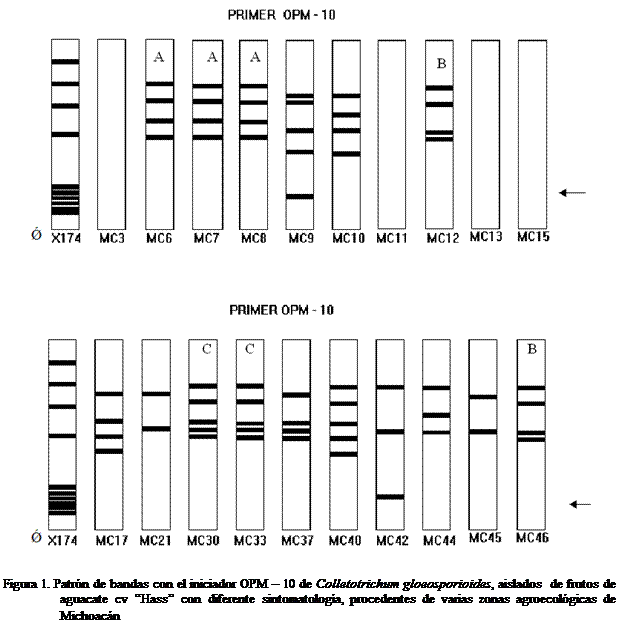
por ser un fragmento demasiado grande. Se puede observar que con el iniciador
OPM-10, los MC 6, 7 y 8 presentan patrones idénticos, los tres monoconidiales
son de la misma localidad, Toreo el alto de Uruapan, y presentan el mismo
síntoma de la enfermedad (viruela), lo mismo sucede con el 30 y 33 pero con diferente polimorfismo a los primeros, en este caso su origen es
diferente, San Juan Nuevo y Milpillas respectivamente con la misma
manifestación de la enfermedad del tipo antracnosis, los monoconidiales 12 y 46
también son idénticos en su patrón de ADN a pesar de ser de localidades
diferentes, Jujucato y Tingambato pero los síntomas que manifiestan en el fruto
son idénticos (manchas superficiales). El resto de los monoconidiales
amplifican fragmentos de diferente peso molecular característicos a cada uno y
sólo la banda de bajo peso molecular es la misma en MC 9 y 42 (Figura 1).
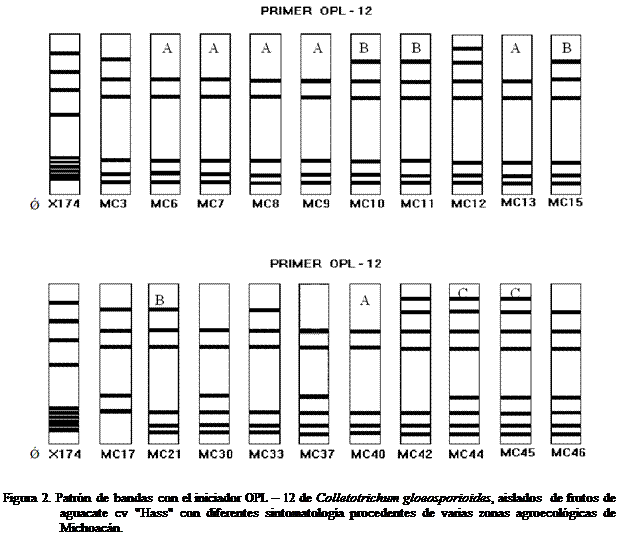
Con respecto al iniciador OPL-12, se revelaron fragmentos iguales en los MC 6, 7, 8, 9, 13 y
40, los tres primeros pertenecen a la misma localidad (Toreo el alto de
Uruapan) y manifiestan el síntoma de viruela, el cuarto procede de la mesa y
produce el mismo síntoma, en cuanto a los dos últimos el primero es del
Tarascon y el segundo de Tanaxuri, ambos producen síntomas de antracnosis; por
otro lado los MC 10, 11, 15 y 21 presentaron un patrón de bandeo igual y
diferentes a los primeros, sin embargo entre ellos no existe ninguna relación
de localidad o síntoma; otros que fueron idénticos entre ellos son el 44 y 45,
ambos presentaron el síntoma de antracnosis en el fruto atacado, el resto
fueron diferentes (Figura 2).
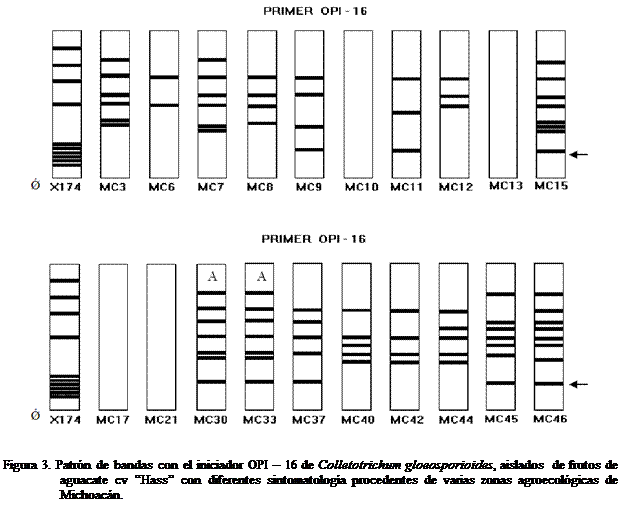
El iniciador OP1-16, reveló un patrón muy heterogéneo para
la mayoría de los MC, a excepción de los MC 30 y 33 que fueron idénticos (con
síntomas de antracnosis), por otro lado se observó que los MC 9, 11, 15, 30,
33, 37, 45, y 46 comparten la característica de presentar una banda de bajo
peso molecular indicada por la flecha en
la Figura 3.
Por otro lado el iniciador OPD-11, reveló una banda de bajo peso
molecular en los MC 6, 7, 8, 10, 11, 21, 37 40, 42, 44, 45 y 46 muy similar que
la revelada por el iniciador OP1-16; se observa también que los únicos
monoconidiales idénticos fueron el MC 7 y 8 que proceden de la misma localidad
presentando ambos el síntoma de viruela en el fruto atacado, finalmente el
resto fueron altamente polimórficos, aunque compartían algunas bandas iguales,
como las mostradas con la doble flecha (←) (Figura 4).

Finalmente el iniciador OPA-16, fue el que mostró la mayor variación en el
patrón de bandas de los monoconidiales, siendo idénticos solamente los MC 33 y
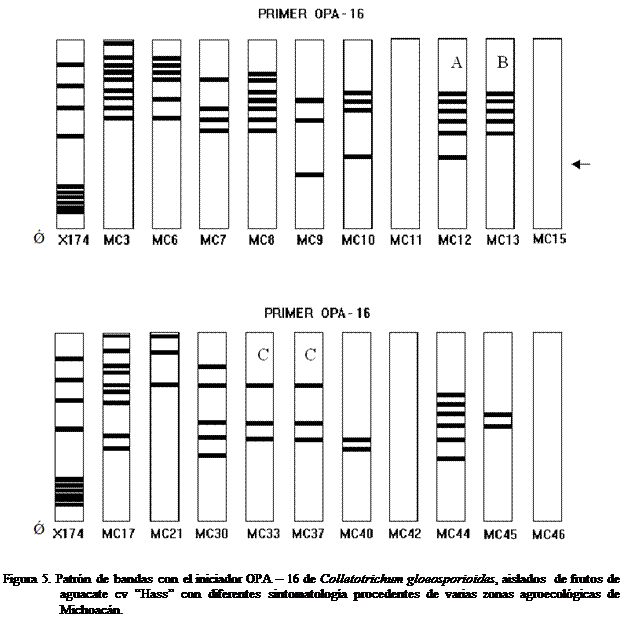
Con las frecuencias de las bandas de cada monoconidial
y tomando en cuenta todos los iniciadores se calcularon las distancias
genéticas, obteniéndose una matriz general, donde se observan las distancias
entre los monoconidiales debida al
polimorfismo de las bandas de ADN
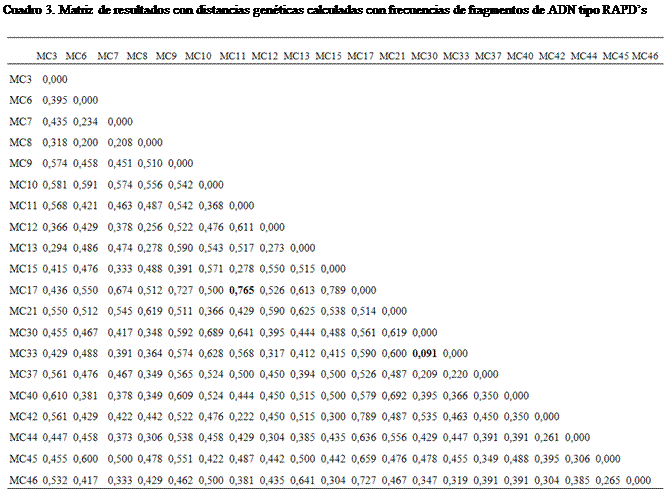
presentadas. Se encontró que la distancia más pequeña o cercanía genética más
estrecha está entre los monoconidiales MC30 y MC33 (0.091), procedentes de la
región de Uruapan, el primero con síntomas de antracnosis y el segundo con
síntomas de antracnosis y coarteadura. La distancias más grandes se presentaron
entre los monoconidiales MC15 y MC17 (0.785) ambos procedentes de la región de
Tacámbaro de la localidad de Longaniza y
En la matriz del análisis de Cluster con
agrupamiento completo se encontró una desviación estándar del total de la
muestra de 0.1486 (Cuadro 4), lográndose un dendrograma con dos grupos
principales, uno con un solo individuo, el cual genéticamente presenta
características completamente diferentes al resto. En el grupo donde está el resto
de los monoconidiales se subdivide a su vez en dos subgrupos los cuales
presentan una segunda subdivisión, que nos permite realizar una cierta
clasificación de los monoconidiales estudiados mediante el polimorfismo de su
ácido desoxirribonucleico, amplificado al azar (Figura 6).
|
Cuadro
4. Valores propios y proporción de variación explicada en el análisis
multivariado de Cluster con agrupamiento completo de 20 monoconidiales de Colletotrichum gloeosporioides aislados
de frutos de aguacate cv Hass procedentes de Tacámbaro y Uruapan, Michoacán,
México y caracterizados por fragmentos de ADN amplificado al azar (RAPD´s). |
|||
|
Monoconidiales |
Valor propio |
Proporción de la Varianza |
|
|
Explicada |
Acumulativo |
||
|
1 |
0,128201 |
0,290208 |
0,29021 |
|
2 |
0,115560 |
0,261593 |
0,55180 |
|
3 |
0,055326 |
0,125242 |
0,67704 |
|
4 |
0,039090 |
0,088487 |
0,76553 |
|
5 |
0,030726 |
0,069555 |
0,83508 |
|
6 |
0,018485 |
0,041845 |
0,87693 |
|
7 |
0,016000 |
0,036220 |
0,91315 |
|
8 |
0,012620 |
0,028567 |
0,94172 |
|
9 |
0,007487 |
0,016949 |
0,95867 |
|
10 |
0,006375 |
0,014430 |
0,97310 |
|
11 |
0,003689 |
0,008351 |
0,98145 |
|
12 |
0,002895 |
0,006553 |
0,98800 |
|
13 |
0,001633 |
0,003697 |
0,99170 |
|
14 |
0,001528 |
0,003459 |
0,99516 |
|
15 |
0,000721 |
0,001632 |
0,99679 |
|
16 |
0,000580 |
0,001313 |
0,99810 |
|
17 |
0,000479 |
0,001085 |
0,99919 |
|
18 |
0,000249 |
0,000564 |
0,99975 |
|
19 |
0,000110 |
0,000249 |
1,00000 |
|
20 |
-0,000000 |
-0,000000 |
1,00000 |
|
Desviación
estándar del total de la muestra =
0,14862 Distancia
media entre observaciones = 0,912096 |
|||
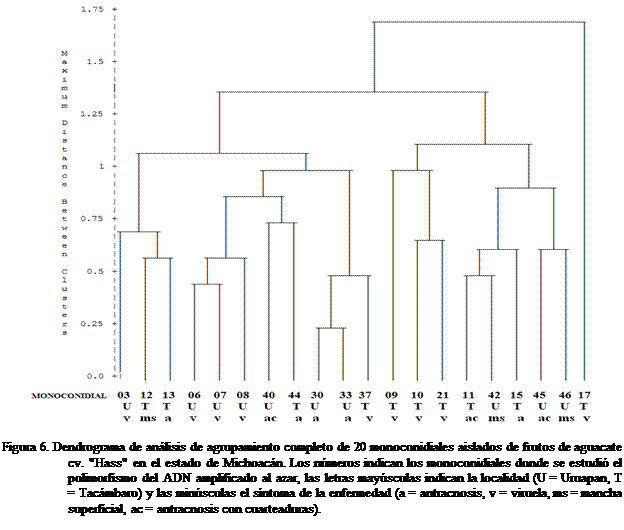
En el dendrograma
se observa la formación de dos grupos principales uno con el monoconidial 17 y
el segundo con el resto, el cual se
divide en dos subgrupos en él más pequeño de ellos tenemos predominantemente
los monoconidiales de Tacámbaro con síntoma de viruela y en el otro aparecen
los de Uruapan con el mismo síntoma. A pesar de que el síntoma es el mismo la
conformación genética de los monoconidiales de una y otra localidad difieren,
como lo indica el agrupamiento de los mismos en el dendograma (Figura 6). En
general se observa una tendencia de agrupamientos por localidad y por síntomas
de la enfermedad. Con respecto a los resultados de agrupamiento por localidades
concuerda con los obtenidos por Mills et
al., (1992). Estos resultados nos
permiten de manera preliminar obtener marcadores genéticos cuya expresión
fenótipica nos apoyen a agrupar variantes de C. gloeosporioides que pudieran influir en el manejo de la
enfermedad.
Los valores que
explican la varianza que representa cada uno de los componentes elaborados
mediante los datos de distancias genéticas obtenidos a partir de la frecuencia
de fragmentos de ADN de diferente longitud característicos para cada
monoconidial se muestran en el cuadro 5. Los tres primeros componentes explican
el 65.7 % de la variación debida a la composición genética de los
monoconidiales estudiados mediante marcadores RAPD’s. La distribución genética
en el espacio de cada uno de los monoconidiales con base a sus distancias
genéticas se muestra en la figura 7,
donde se observa claramente las distancias genéticas más grandes entre los monoconidiales
15, 11, 42 y 46 (0.789, 0.765, 0.636 y 0.727 respectivamente). Se observa que
el monoconidial 17 se encuentra al extremo derecho de la gráfica. Esta
tendencia también se observó en el dendrograma donde el monoconidial 17 quedo
aislado del resto, esto pudiera tener alguna explicación relacionada con una
posible mutación de su genoma, lo cual debería de comprobarse en estudios
posteriores.
|
Cuadro 5. Valores propios y proporción de la
variación explicada por los Componentes Principales de 20 monoconidiales de Colletotrichum gloeosporioides caracterizados
por fragmentos de ADN amplificado al
azar (RAPDs) |
|||
|
Componente Principal |
Valor propio |
Proporción de la varianza |
|
|
Explicada |
Acumulada |
||
|
CPRIN1 |
5,74709 |
0,287354 |
0,28735 |
|
CPRIN2 |
4,80019 |
0,240009 |
0,52736 |
|
CPRIN3 |
2,61100 |
0,130550 |
0,65791 |
|
CPRIN4 |
1,91867 |
0,095933 |
0,75385 |
|
CPRIN5 |
1,44132 |
0,072066 |
0,82591 |
|
CPRIN6 |
0,86479 |
0,043240 |
0,86915 |
|
CPRIN7 |
0,78288 |
0,039144 |
0,90830 |
|
CPRIN8 |
0,59752 |
0,029876 |
0,93817 |
|
CPRIN9 |
0,37105 |
0,018553 |
0,95672 |
|
CPRIN10 |
0,31164 |
0,015582 |
0,97231 |
|
CPRIN11 |
0,18151 |
0,009076 |
0,98138 |
|
CPRIN12 |
0,13375 |
0,006688 |
0,98807 |
|
CPRIN13 |
0,07341 |
0,003671 |
0,99174 |
|
CPRIN14 |
0,06865 |
0,003432 |
0,99517 |
|
CPRIN15 |
0,03268 |
0,001634 |
0,99681 |
|
CPRIN16 |
0,02781 |
0,001390 |
0,99820 |
|
CPRIN17 |
0,02047 |
0,001024 |
0,99922 |
|
CPRIN18 |
0,01100 |
0,000550 |
0,99977 |
|
CPRIN19 |
0,00458 |
0,000229 |
1,00000 |
|
CPRIN20 |
0,00000 |
0,000000 |
1,00000 |
En relación a su
origen geográfico y síntomas que producen los monoconidiales al infectar frutos
de aguacate cv. Hass, en la Figura 7 se observa que los genotipos de los monoconidiales 8, 6 y 3 cuyos síntomas
es de viruela y provienen de Uruapan se agrupan hacia la derecha. Así mismo, se
forma otro grupo en el centro bajo con
los monoconidiales 37, 21, 10 y 9 procedentes de Tacámbaro también con síntoma
de viruela. A pesar de que ambos grupos producen el mismo síntoma en el fruto
infectado, la composición genética de los grupos es diferente. Esto debido
probablemente a las diferentes presiones ambientales y de manejo a que esta
sometido C. gloeosporioides en las
localidades, debido a que el municipio de Uruapan fue el primero donde se
introdujo el aguacate cv. Hass, por lo tanto; el control químico se ha
usado durante más tiempo, en contraste
con Tacámbaro que es un municipio relativamente
"joven", lo cual pudiera haber influido en mutaciones del
hongo. En la parte central alta se
observa un grupo formado con los monoconidiales 40, 33 y 30 cuya característica
en cuanto a síntomas es de antracnosis y
su origen es Uruapan. Por otro lado, en el extremo izquierdo se concentran los
monoconidiales 44, 15 y 11 con el mismo síntoma pero provenientes de Tacámbaro,
ambiente que determina la composición genética de este grupo en particular.
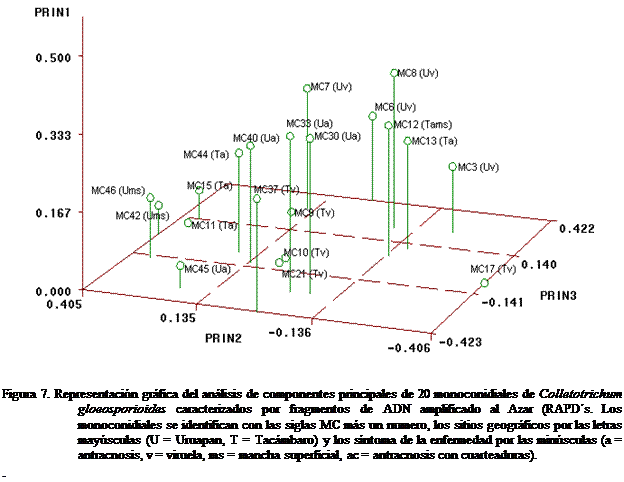
Finalmente en la
parte mas alejada de este extremo tenemos los monoconidiales 46 y 42, con
síntomas de manchas superficiales ambos de Uruapan, distantes pero
genéticamente muy cercanos entre sí. Algunos monoconidiales como el 45,7, 12,
13 y 17 escapan a las tendencias señaladas, esto se debe a la necesidad de
incrementar el número de iniciadores en
las pruebas de amplificación de ADN, para lograr mayor precisión en la caracterización genética de los
monoconidiales en estudio.
El índice de variabilidad genética arrojó un valor de
0.4101 el cual es muy alto incluso cuando se compara con plantas superiores
como el tomate de cáscara silvestre que tiene un índice de 0.3828 y maíz
criollo con un valor de 0.3500 (Montero et
al. 1998). Esta variabilidad genética tan amplia puede ser debida a la gran
patogenicidad y al amplio rango de hospedantes del patógeno observado en la
naturaleza. La amplia variabilidad
genética encontrada concuerda con los
reportado por Sutton (1992), así como lo reportado por Braithawaite (1990) mediante el análisis del
ADN mitocondrial y nuclear usando la técnica de RFLP con aislamientos de C. gloeosporioides pero de diferente
hospedante. Bailey y Jeger, (1992) reportaron una alta variación en
aislamientos procedentes del mismo
cultivo y del mismo país, en contraste, encontraron similitud en aislamientos
procedentes de diferente cultivo (papayo y
aguacate).
CONCLUSIONES
- Las poblaciones de Colletotrichum son diferentes y los monoconidiales se separan
de acuerdo a su lugar de procedencia y a síntomas.
- La variabilidad genética es muy amplia, lo cual se
refleja en el alto valor del índice de variabilidad genética que presentan
los monoconidiales.
- De los iniciadores ensayados OPA-16 y OPM-10 son los más
eficientes para caracterizar las poblaciones de C. gloeosporioides.
LITERATURA CITADA
Armstrong, J.; A. Gibb, R. Peakall and G.
Weiller. 1995. The rapid distance package version 104. Australian National
University, Camberra, Australia. Sitio internet http://life.anu.edu. Au/molecular/software/rapd.html.
Bailey,
A. J. and J. M. Jeger. 1992. Colletotrichum:
biology, pathology and control. British
Society for Plant Pathology. C.A.B International. 388 p.
Braithwaite,
K. S.; J. A. Irwin and J. M. Manners. 1990. Restriction fragment length
polymorphisms in Colletotrichum
gloeosporioides infecting Stylosanthes
spp. in Australia. Mycological Research
94: 1129-1137.
Hu,
J. and C. F. Quiros. 1991. Identification of broccoli and cauliflower cultivars
with RAPD markers. Plant Cell Rep. 10: 505-511.
Kolmer,
J. A. 1991. Evolution of distintict populations of Puccinia recondita f. sp tritici in Canadá Phytopathology 81:
316-322.
Lázaro, B. V. 1985. Incidencia de la
antracnosis (Colletotrichum
gloeosporioides, Penz ) en aguacate
variedad Hass almacenado en refrigeración
y su relación con el manejo
cultural de las huertas en Uruapan,
Mich. Tesis. Ingeniero Agrícola UNAM
.Cuautitlán Izcalli, Estado de México. 75 p.
Mills,
P. R.; S. Sreenivasaprasad and A. E. Brown. 1992. Detection and differentiation
of Colletotrichum gloeosporioides isolates
using PCR. Microbiology 98: 137-144.
McDonald,
B. A. and J. M. McDermott. 1993. Population genetics of plant pathogenic fungi.
Electrophoretic markers give unprecedented precision to analyses of genetic
structure of populations. BioScience 43:5: 311-319.
Montero,
T. V.; H. L. Montalvo, J. Morales
G., R. H. Azpiroz, L. A. Peña y M. A.
Gálvez. 1998. Estimación de la
variabilidad genética intrapoblacional mediante el uso de fragmentos de ADN
amplificados al azar. Agric. Tec. México 24:83-89.
Morales G., J. L. 1996. Caracterización
cultural, morfologica, patogenica y molecular de Colletrotrichum gloeosporioides
Penz. causante de la antracnosis del aguacate en Michoacán. Tesis de Maestría
Colegio de Postgraduados, Montecillos,
México 96 p.
Sreenivasaprasad, S.; A. E. Brown and R.
P. Mills. 1992. DNA sequence variation and interrelationships among Colletotrichum species causing
strawberry anthracnose. Phisiol. Mol. Plant Phatol. 41: 265-281.
Sutton,
B. C. 1992. In: Bailey, A.J. and Jeger, J.M. 1992. Colletotrichum: biology, pathology and control. British Society for Plant Pathology. C.A.B
International. 388 p.
Téliz, O. D. 1999. El aguacate y su
manejo integrado. Mundi prensa México, S.A. de C.V. 215 p.
United
States Department of Agriculture (USDA). 1997. Docket No. 94-116-5. Final rule:
Hass avocados from Mexico. Federal Register USA. 102 p.
Williams,
J. G. K.; A. E. Kubelik, K. L. Levak and S. C. Tingey. 1990 DNA polimorphisms
amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research 18: 6531-6535.
Página diseñada por Prof. Jesús Rafael
Méndez Natera
TABLA
DE CONTENIDO DE LA REVISTA CIENTÍFICA UDO AGRÍCOLA



